데이타 불러오기
다음과 같은 패키지를 먼저 불러옵니다.
library(PKNCA) # devtools::install_github('billdenney/PKNCA')
library(tidyverse) # devtools::install_github('tidyverse/tidyverse')
library(knitr) # for kable먼저 Theoph 데이타를 살짝 손보고자 합니다. Tibble형태로 바꾸고 factor화된 Subject 열을 숫자로 바꾸고 정렬할 것입니다.
rawTheoph <- datasets::Theoph %>%
as_tibble() %>%
mutate_at(vars(Subject), as.numeric) %>%
arrange(Subject, Time)
rawTheoph %>% head %>% kable| Subject | Wt | Dose | Time | conc |
|---|---|---|---|---|
| 1 | 80 | 4 | 0.00 | 0.00 |
| 1 | 80 | 4 | 0.27 | 1.29 |
| 1 | 80 | 4 | 0.58 | 3.08 |
| 1 | 80 | 4 | 1.15 | 6.44 |
| 1 | 80 | 4 | 2.03 | 6.32 |
| 1 | 80 | 4 | 3.57 | 5.53 |
농도 데이타를 concTheoph에 넣고, 투약 데이타를 doseTheoph에 넣었습니다.
concTheoph <- PKNCAconc(rawTheoph, conc~Time|Subject)
concTheoph## Formula for concentration:
## conc ~ Time | Subject
## With 12 subjects defined in the 'Subject' column.
## Nominal time column is not specified.
##
## First 6 rows of concentration data:
## Subject Wt Dose Time conc exclude
## 1 80 4 0.00 0.00 <NA>
## 1 80 4 0.27 1.29 <NA>
## 1 80 4 0.58 3.08 <NA>
## 1 80 4 1.15 6.44 <NA>
## 1 80 4 2.03 6.32 <NA>
## 1 80 4 3.57 5.53 <NA>## Dosing data needs to only have one row per dose, so subset for that first.
doseRawTheoph <- rawTheoph %>%
filter(Time == 0) %>%
select(Subject, Dose, Time)
doseRawTheoph %>% head %>% kable| Subject | Dose | Time |
|---|---|---|
| 1 | 4.00 | 0 |
| 2 | 4.95 | 0 |
| 3 | 4.53 | 0 |
| 4 | 4.92 | 0 |
| 5 | 4.53 | 0 |
| 6 | 4.40 | 0 |
doseTheoph <- PKNCAdose(doseRawTheoph, Dose~Time|Subject)## Assuming route of administration is extravascular## Assuming instant dosing (duration=0)doseTheoph## Formula for dosing:
## Dose ~ Time | Subject
## Nominal time column is not specified.
##
## First 6 rows of dosing data:
## Subject Dose Time exclude route duration
## 1 4.00 0 <NA> extravascular 0
## 2 4.95 0 <NA> extravascular 0
## 3 4.53 0 <NA> extravascular 0
## 4 4.92 0 <NA> extravascular 0
## 5 4.53 0 <NA> extravascular 0
## 6 4.40 0 <NA> extravascular 0그림 그리기
concTheoph는 plot으로 볼 수 있습니다.
plot(concTheoph)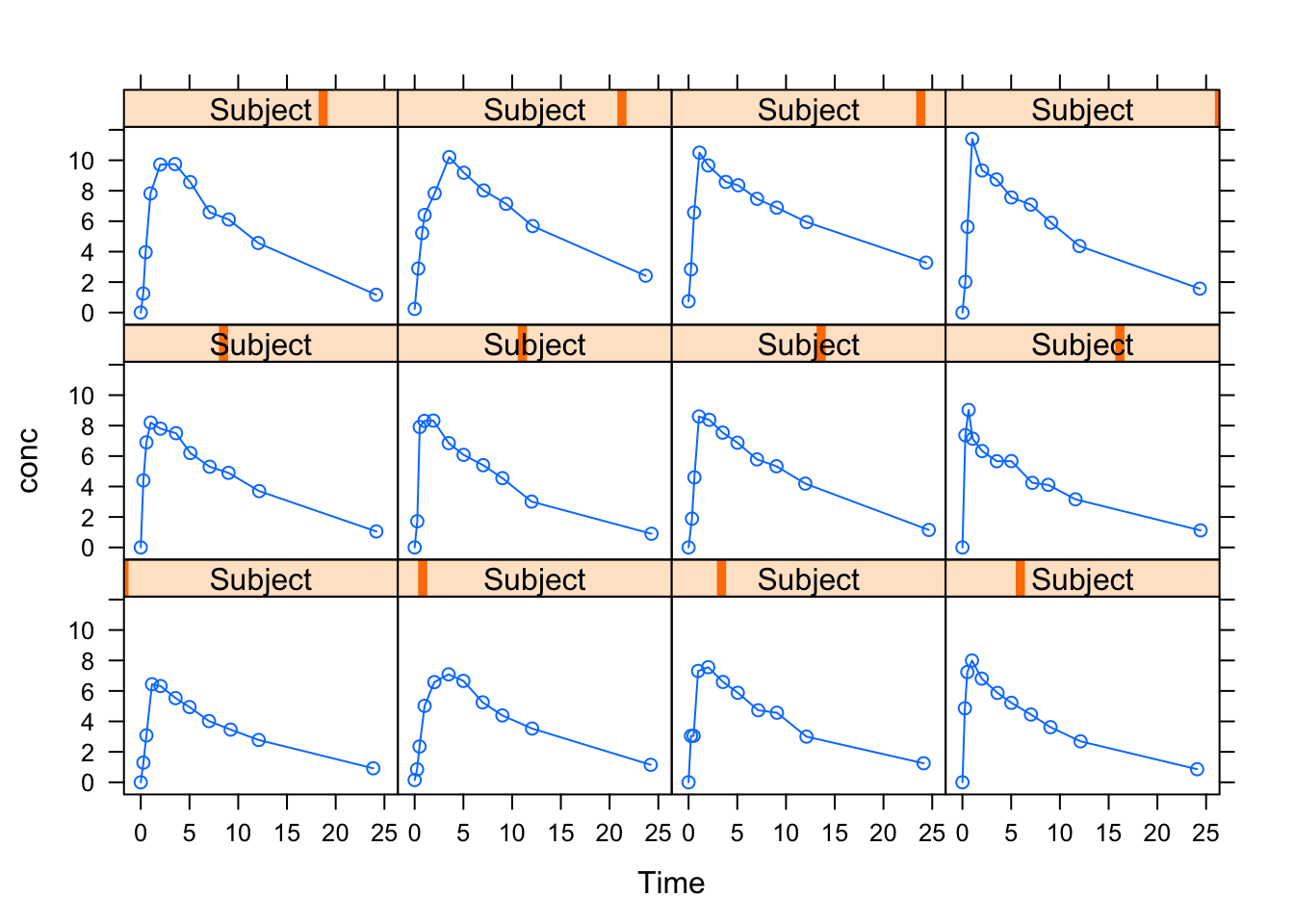
농도와 투약 합치기
농도와 투약 정보를 합치는 것은 PKNCAdata 함수를 사용합니다.
dataTheoph <- PKNCAdata(concTheoph, doseTheoph)계산하기
파라메터는 pk.nca 함수를 통해 구할 수 있습니다.
ncaTheoph <- pk.nca(dataTheoph)
ncaTheoph$result %>% head %>% kable| start | end | Subject | PPTESTCD | PPORRES | exclude |
|---|---|---|---|---|---|
| 0 | 24 | 1 | auclast | 71.6970150 | NA |
| 0 | Inf | 1 | cmax | 6.4400000 | NA |
| 0 | Inf | 1 | tmax | 1.1500000 | NA |
| 0 | Inf | 1 | tlast | 23.8500000 | NA |
| 0 | Inf | 1 | clast.obs | 0.9200000 | NA |
| 0 | Inf | 1 | lambda.z | 0.0877957 | NA |
summary(ncaTheoph) %>% kable| start | end | N | auclast | cmax | tmax | half.life | aucinf.obs |
|---|---|---|---|---|---|---|---|
| 0 | 24 | 12 | 74.6 [24.3] | . | . | . | . |
| 0 | Inf | 12 | . | 8.65 [17.0] | 1.14 [0.630, 3.55] | 8.18 [2.12] | 115 [28.4] |
이상으로 PKNCA 패키지에 대해 간략히 살펴보았습니다.
References
Denney, Bill, and Clare Buckeridge. 2017. PKNCA: Perform Pharmacokinetic Non-Compartmental Analysis. https://CRAN.R-project.org/package=PKNCA.